matplotlibのsubplotを使った図の作成
matplotlibの使い方、subplot関数の使い方についてまとめました
基本準備
Irisのデータを使う
import numpy as np import pandas as pd import matplotlib.pyplot as plt df = sns.load_dataset("iris")
通常のグラフ作成
軸の範囲・表示値設定
- xlim:x軸の値範囲
- ylim:y軸の値範囲
- xticks:表示するメモリの値
軸のテキスト設定
- xlabel:x軸のラベル設定
- ylabel:y軸のラベル設定
- title:グラフのタイトル設定
plt.plot(df.sepal_width, df.sepal_length, '.', label="test") plt.xlim(2.0,5.0) plt.xticks([2,3,4, 5]) #メモリの設定 plt.yticks([4,5,6,7, 8]) plt.xlabel('sepal_width')# x軸のラベル設定 plt.ylabel('sepal_length') plt.title('test',fontsize=20) plt.legend(loc='best')# 一番いい感じのところで設定してくれる

重ねてグラフ表示
まずは普通に表示する
plt.plot(df.sepal_width, df.sepal_length,'.',color='red') plt.plot(df.sepal_width, df.petal_length,'x',color='blue')

だけど、違う種類同士のグラフだとy軸が重なり合って、良く分からない感じの図になる。
plt.hist(df.sepal_width , bins=20) plt.plot(df.sepal_width, df.sepal_length,'.',color='red')
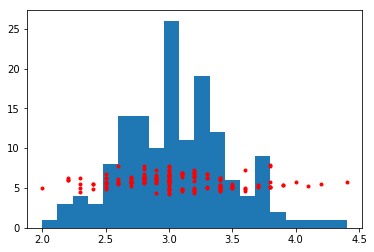
解決策その1:別々のグラフにして表示する
まずfigureのインスタンスと言う箱的なのを作る。
その後、箱の中をサブプロットと言う関数で分けて、別々のグラフをその中で表示させる。

fig = plt.figure(figsize=(8,8)) ax1 = fig.add_subplot(211) ax2 = fig.add_subplot((212), sharex=ax1)#y軸の共有 ax1.plot(df.sepal_width, df.sepal_length,'.',color='red') ax2.hist(df.sepal_width , bins=20)
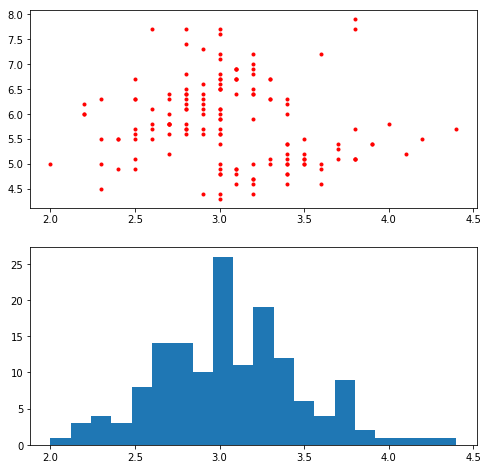
解決策その2:グラフを上下に表示させる
ザックリとやってることをいかにまとめた。

fig = plt.figure(figsize=(6,6)) # サブプロットを8:2で分割 ax1 = fig.add_axes((0, 0.2, 1, 0.8))#[left(左端からの距離), bottom(下からの距離), width(幅), height(高さ)] ax2 = fig.add_axes((0, 0, 1, 0.2), sharex=ax1) # 散布図のx軸のラベルとヒストグラムのy軸のラベルを非表示 ax1.tick_params(labelbottom="off") #ラベル設定 ax1.set_ylabel('sepal_length') ax2.set_ylabel('count') ax1.plot(df.sepal_width, df.sepal_length,'.',color='red') ax2.hist(df.sepal_width , bins=20)
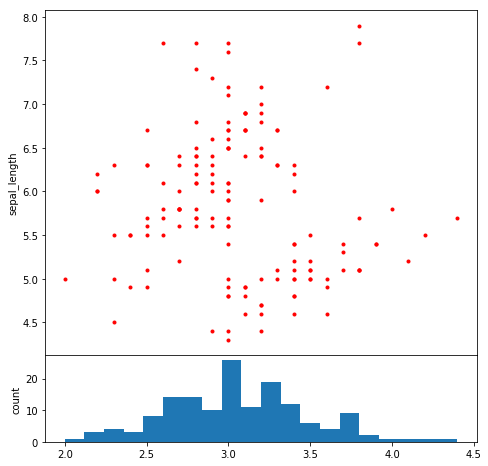
参考資料
http://bicycle1885.hatenablog.com/entry/2014/02/14/023734
解決策その3:左右のy軸で別々の値を表示させる
Figureのインスタンスを生成し、Axesのインスタンスの中でx軸を共有させてグラフを作るといい感じに作れる。(twinx()関数)
詳しくは後でまとめるので、一旦コードだけ。
fig = plt.figure(figsize=(6,6)) # サブプロットを作成・x軸の共有 ax1 = fig.add_subplot(111) ax2 = ax1.twinx() #ラベル設定 ax1.set_xlabel('sepal_width') ax1.set_ylabel('sepal_length') ax2.set_ylabel('count') ax1.plot(df.sepal_width, df.sepal_length,'.',color='red') ax2.hist(df.sepal_width , bins=20,alpha=0.4)

補足
- legendについて
legendを表示させる場合は、普通にやると上手くいかない。
なので、以下のようにして一つにまとめてやる必要がある。
handler1, label1 = ax1.get_legend_handles_labels() handler2, label2 = ax2.get_legend_handles_labels() ax1.legend(handler1 + handler2, label1 + label2, loc='best',fontsize=14)
- axesでのx軸目盛について
こちらも普通にax1.xtickとかやっても上手くいかない。
ax1.set_xticklabels([~~], rotation = 90)とかで処理できる
「生命情報向けの機械学習」を触ってみた
「生命情報向けの機械学習入門」という内容で、生物×機械学習という分野でコンテンツを作られている方のgitを見つけて色々触ってみたので、書き留めておきます。
「4章 : 配列を解析する深層学習」という内容を触ってみました。 内容としては、以下のようなものです。
世界のChIP-seqデータ(転写因子結合サイト実験)を収集し解析したデータベースであるChIP-Atlas (Oki et al. EMBO reports 2018) から、一次解析の終了したChIP-seqデータを基に、転写因子結合の有無を予測します。
一言でいうと、配列から転写因子CTCFの結合を予測しています。 100塩基の配列をCNNを使って学習・予測を行わせています。
前回紹介した論文でも配列をニューラルネットワークを使って学習させていたのですが、 nissyl.hatenablog.com
個人的にブースティング系の方が精度でるし、その後の考察もやりやすいんじゃないのかなとずっと思っていたので、試しにLightGBMで学習させてみました。
前処理・学習
とりあえず、データを拝借し多次元配列で作られていたのを、1次元に戻して配列をダミー化しました。
 1がアデニン・2がシトシン・3がグアニンです(チミンは次元削除されました)
1がアデニン・2がシトシン・3がグアニンです(チミンは次元削除されました)
こんな雑な処理でいいのかな(?)と思いますが、
まぁ気にせず、一旦パラメーターチューニングせずに、最後まで一気にいきました。
(一応カテゴリ変数の取り扱いについて記述したことがあるので、きになる方はこちらどうぞ)
df_train = df.copy() #変数を配列へ変換 import lightgbm as lgb from sklearn import metrics from sklearn import datasets from sklearn.model_selection import train_test_split X = df_train.drop("objective_variable" , axis=1).values y = df_train["objective_variable"].values X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=1, stratify=y) # とりあえず適当にやってみる lgbm_params = { 'task' : 'train', 'boosting_type' : 'gbdt', 'objective' : 'binary',#2値分類 'metric' : {'auc'}, 'num_leaves' : 31, 'learning_rate' : 0.05, 'feature_fraction' : 0.9, 'bagging_fraction' : 0.8, 'bagging_freq': 5, 'verbose' : 10 } columns = (df_train.columns[:300]).tolist() model = lgb.train(lgbm_params, lgb_train,num_boost_round=2500, valid_sets=lgb_eval,feature_name=columns)
評価
とりあえず、AUC, Log lossと、一応accuracyを見ました。
[in] y_pred = model.predict(X_test, num_iteration=model.best_iteration) predictions = [round(value) for value in y_pred] print('roc-auc: %.4f' %mtr.roc_auc_score(y_test, y_pred)) print('log-loss: %.4f' %mtr.log_loss(y_test, y_pred)) print('accuracy: %.4f' %accuracy_score(y_test, predictions) [out] roc-auc: 0.9443 log-loss: 0.4966 accuracy: 0.8451
AUCはいい感じです。
図も書いてみました。


ただ若干、log-lossの値がでかいので基礎集計をみてみます。
[in] pd.Series(y_pred).describe() [out] count 32682.000000 mean 0.499419 std 0.150936 min 0.166480 25% 0.360993 50% 0.499363 75% 0.638689 max 0.845363
やっぱり、予測スコアは全体的に0.5付近に集中してそうですね。
まぁ精度を求めても仕方ないので、次に特徴量の評価をみてみます。
最近書いたこれです。
imortance_typeはgainをみています。
lgb.plot_importance(model, figsize=(14, 6), importance_type='gain', max_num_features=10)

使うアルゴリズムやimortance_typeによっても変わってくるので、一概には言えないですが
78番目のグアニンと15番目のアデニンが、何かしら結合に効いてるぽいです。
まとめ
配列系の予測も、ブースティング系のアルゴリズムを使っていけるんじゃないかなと思いました。
多分、ニューラルネットワークと併せて最後にブレンディングする感じですかね。
塩基評価のところは、もっと本気で色んな観点から見ないと、本当は何も言えないので、あてにしないでください。
最後に、勝手にデータを使わせて頂いてありがとうございました!!
カテゴリ変数の取り扱い方法
都道府県の名称など、値自体に意味が無い変数を扱う場合は、そのまま機械学習にかけてしまうと意味不明な結果が出る。

ダミー変数化
変数を各種類ごとのカラムに分けて、0,1表記にしよう。と言うもの
一番ポピュラーなカテゴリ変数の扱い。

ターゲットエンコーダー
各カテゴリ変数の各値で、正解である割合を各変数の持つ変数値として持たせる方法

参考資料 Feature Engineering
somoothing_encoder
図 3の「埼玉」みたいに、ターゲットエンコーダーを実際の現場で活用すると、たまたま少ない数の目的変数が全て1であった場合、ターゲットエンコーダーの値が1になって信頼性が疑わしい。
ので、パラメーターとして、学習データの中の「埼玉の割合」を重み付けする係数を追加する。
式は以下の感じ。

これを通じて、目的変数の発生確率を重み付け、たまたま「埼玉」が1になる時に、信頼性を重み付けられるようにしている。
詳しくは参考資料を
A Preprocessing Scheme for High-Cardinality Categorical Attributes in Classification and Prediction Problems
light gbmのカテゴリ変数の扱い
種類がめっちゃ多いカテゴリ変数を、ダミー化して変数を構築すると、カテゴリ変数の種類の分だけ列が増え計算量が増大してしまうし、最適化がされにくいらしい。
Particularly for high-cardinality categorical features, a tree built on one-hot features tends to be unbalanced(ドキュメントより引用)
とりあえず、カテゴリ変数を、各分岐点での (sum_gradient / sum_hessian)の値でヒストグラムを構築し、損失関数が最小になるところで分岐させる
documentによると、大体はダミー化するよりいいモデルができるらしい。
CatBoost
上記に書いたみたいに、lightGBMでは分岐させるときに、データの勾配を使って学習を行わせる。 ただ、これだと真のデータ分布に従うか分からないのに、観測データだけでモデルを作るようなものなので、バイアスが掛かって過学習してしまう。 (ちょっといい感じに日本語に訳せないので、論文の原文を引用。)
This leads to a shift of the distribution of estimated gradients in any domain of feature space in comparison with the true distribution of gradients in this domain, which leads to overfitting.
解決策としては、勾配の分布をモデリングするモデルを別途作りましょう。と言う感じ。 カテゴリ変数を扱う上では有用らしいが、全体の精度としては正直LightGBMとかと変わらない気がする。
とりあえず、CatBoostではカテゴリ変数を数値に変換しており、方法は以下の式のようになる。
![]()
- CountInclass :ターゲットエンコーダーと同じ
- propr:ユーザー側で指定するパラメーター
- totalCount:該当の値の数
参考資料
CatBoost: gradient boosting with categorical features
support
Transforming categorical features to numerical features
機械学習を活用した変数重要度評価
モデルを作った後、説明する際に
「ここら辺の特徴量が効いてます。」 的なことを言わないといけない。
そこで、変数重要度をよく使う。
ランダムフォレストやXGboostでの変数の重要度評価をメモがてらまとめてみた。
RandomForestを活用した変数重要度評価
RandomForestについての整理
RandomForestを使った変数評価
変数の重要度は、分岐に使用する変数を入れ替えた際、ジニ係数にどれくらい影響があるかを見ている。(多分)
特徴量が出てくる回数とかではないので、多重共線性があっても重要度は変化しない。
要は、「多重共線性が存在しても、変数間の重要度の順位関係は保存される。」ということ。
そこらへんの話は、この論文に詳しく載ってる。
Variable selection using random forests
XGboostを活用した変数重要度評価
XGboostについての整理
ジニ係数を小さくさせて学習するRandomForestとは違い、Xgboostは損失関数を小さくする向きに学習を進める。
XGboostを使った重要度
XGboostの重要度の定義には、weight, gain, cover の3つがある
- weight:全学習木で、特徴量が分岐に使用された回数
gain:Nodeでの以下の式で定義付けられる値の減少度の平均
 gradientとhessianの意味が、ちょっと何言ってるか分かんない場合は、
とりま損失関数を最小化させる方向に寄与する特徴量を、いい感じに評価してる程度に理解しておけば良いかと。
gradientとhessianの意味が、ちょっと何言ってるか分かんない場合は、
とりま損失関数を最小化させる方向に寄与する特徴量を、いい感じに評価してる程度に理解しておけば良いかと。
たぶん、RandomForestのジニ係数での評価に近い概念
参考にした文章: Introduction to Boosted Treescover:各Nodeにおいて、変数を使って分けたデータの数 この指標の定義が、どこを探しても見つからなかったので感覚での記述。
多分、学習をさせる中でどれくらいのデータを識別するのに特徴量が使われたかの指標のこと。
おそらく、以下のイメージ。(間違ってたらごめんなさい。)

ここだと、特徴量1がcover 100, 特徴量2がcover 50っていう理解。 あとは、全体の集計・平均すれば出る。
一応、このQAを参考。 https://datascience.stackexchange.com/questions/12318/how-do-i-interpret-the-output-of-xgboost-importance
pandasでDataFrameを表示させるときに省略させない方法
何もしないでDataFrameを表示させると、カラムが省略されたり行が省略されたりする。 メモ書きに、DataFrameを省略させない方法について記載する。
カラムを省略させない方法
以下のコードをDataFrameを表示させる前に書いておけばいい。
pd.set_option('display.max_columns', 300)
コード内の300は表示させる最大のカラム数。もっと大きくすれば、表示するカラム数を上げることができる。
行を省略させない方法
カラムのときと同様、以下のコードをDataFrameを表示させる前に書いておけばいい。
pd.set_option('display.max_rows', 300)
こちらも300の部分をいじれば、表示させられる行数を増やすことができる。
たまに使いたくなるけど、中々思い出せないので一応メモ。
Seabornによるグラフの作成 ~相関関係の可視化~
前回に引き続き、seabornの中の関数について、自分のメモがてら紹介していきます。 今回は相関関係の可視化を中心に取り扱います。
import seaborn as sns import pandas as pd df = sns.load_dataset("iris") #データ準備 df['test'] = df['petal_length'] > 3.5 df.head()
sepal_length sepal_width petal_length petal_width species test 0 5.1 3.5 1.4 0.2 setosa False 1 4.9 3.0 1.4 0.2 setosa False 2 4.7 3.2 1.3 0.2 setosa False 3 4.6 3.1 1.5 0.2 setosa False 4 5.0 3.6 1.4 0.2 setosa False
jointplot関数
二変数間の相関を見る時に使います。使い方は分かりやすく、相関をみたい2つの変数を引数に入れて、kindに'reg'を指定してあげると見れます。
sns.jointplot(df.sepal_length, df.sepal_width, kind="reg")

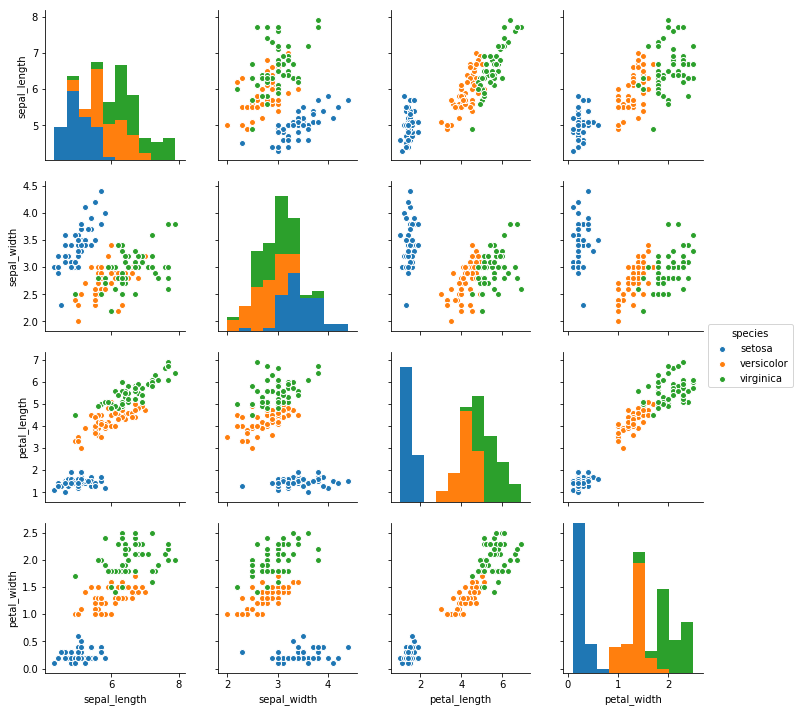
pairplot関数
各変数の散布図を作成することができる。第一引数に元データを入れてあげて、カテゴリごとの違いをみたいなら引数hueに変数を入れてあげる。
sns.pairplot(df, hue="species")
heatmap関数
各変数間の相関の強さをヒートマップにして可視化することができる。 pd.corr()は数値の列のみが対象で、欠損値NaNは除外して算出される。
sns.heatmap(df.corr(), square=True)
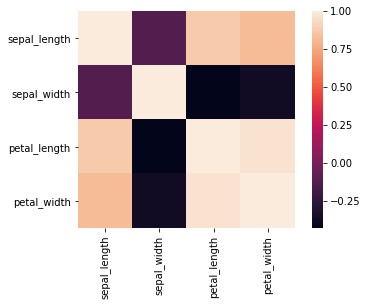
Seabornによるグラフの作成 ~factorplot関数~
グラフを作成する際に、良く使うseabornを紹介します。 seabornは簡単に統計グラフを書くことができる関数を多数提供しています。
ここでは、factorplot関数を使ったグラフ作成を中心に紹介します。
import seaborn as sns df = sns.load_dataset("iris") #データ準備 df['test'] = df['petal_length'] > 3.5 df.head()
sepal_length sepal_width petal_length petal_width species test 0 5.1 3.5 1.4 0.2 setosa False 1 4.9 3.0 1.4 0.2 setosa False 2 4.7 3.2 1.3 0.2 setosa False 3 4.6 3.1 1.5 0.2 setosa False 4 5.0 3.6 1.4 0.2 setosa False
factorplot関数
factorplot関数は、 引数に
- data:使用する元のデータ
- x:x軸に使用するデータ。使用する元のデータのカラム名から選択
- y:y軸に使用するデータ。使用する元のデータのカラム名から選択
- kind:作成するグラフの種類を指定。棒グラフや箱ひげ図など様々なグラフを作成できる
kind = 'bar'にすると以下のような棒グラフが作成できる。
(例) sns.factorplot(data = df, x = 'species', y = 'petal_length', kind = 'bar')

さらに引数hueに、カテゴリデータが格納された列の列名を指定すると、そのカテゴリごとに色分けしたグラフが生成される
(例) sns.factorplot(data = df, x = 'species', y = 'petal_length', hue = 'test', kind = 'bar')

kind = 'box'にすると以下のような箱ひげ図が作成できる。

他にも、kind = 'violin'にするとヴァイオリンプロットができたりするが、自分はあまり使用しないので割愛します。